
Ai真的可以看得懂图像嘛
VQA-MED+Byesian实验记录:代码+方法+实验+调参
我的结论是想让机器以我们所认知的通过记忆推理回答问题这还是一个伪命题
视觉问答(VQA)背景
明白一个本质的问题,一张照片和一个句子对是如果进行向量运算获得最终的预测的
VQA梗概
视觉问答(VQA)是最近几年出现的一个热门研究领域,是一个综合CV视觉推理能力(目标检测,图像分类等)和NLP语言理解能力而形成的一个综合性多学科的任务。比如有一张图片:
四大天王
我想要知道:
图片中有几个人?(开放)
他们是什么职业?(开放)
天气是雨天吗?(封闭)
他们在喝酒吗?(封闭)
这些问题人可以轻松回答,但是未经过训练的机器却不可以。不过我们可以把这个问题分为一系列小任务,比如先理解问题句子包含什么关键词(人,天气等)或者这个问题是开放式问题(open-ended)还是封闭式问题(close-ended)。
开放式问题需要按照问题给出合适答案,这样后续还需要使用目标检测的方法处理图像,比如检测图像中人物的数量。
封闭式问题只需要根据关键词回答是或不是,这样后续就需要将任务变成图像分类任务,比如判断手中是否拿的酒杯。可以看出视觉问答任务需要同时结合CV领域和NLP领域的先进技术。并且随着问题的内容和难度的变化,视觉问答的难度也随之不同。
如今,通用视觉问答技术已经扩展到了多个领域,包括按专业领域划分的知识问答、按视频/图片/表格等不同数据类型的问答等,本质上还是CV、NLP两个领域的信息多模态问题。
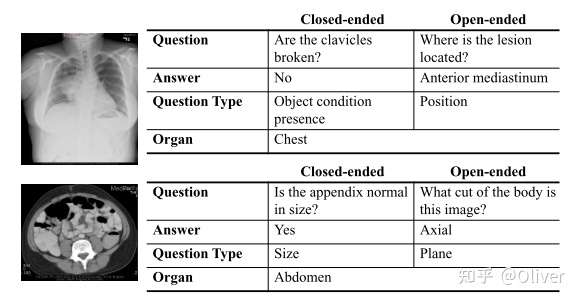
在未来的智慧医疗,AI医疗领域有巨大的应用潜力,能够帮助医生辅助做出病理诊断,甚至自动完成正确的诊断。相比自然图像的视觉问答,医疗图像内容更加单一,问题的复杂度以及推理程度都不如自然图像大。但是医疗图像也非常具有挑战性:一方面,需要保证较高的预测精准度,因为在实际应用场景中,预测结果精度关乎人的生命和健康。另一方面,获取专业医生标注好的医疗图像数据集和问答对(question-answer pair)非常困难,医疗视觉问答数据集通常很小。而那些成功的深度学习模型都含有大量训练参数,在小数据量时很难训练到较好性能(容易过拟合)。
为解决这个问题,最开始是利用迁移学习的方法,让这些大模型在自然图像大数据集上预训练后,再用目标医疗数据进行微调。但由于自然图像和医疗图像的特征之间具有较大的差异,导致迁移后的模型效果并不是很好。接下来介绍的两篇文章就是针对这个问题通过不同的技巧来提升迁移后模型的性能。
VQA技术模型迭代
传统模型:Vencoder+Qencoder+fusing algorithm+answering component
这是VQA问题最经典也最主流的结构,多模态体现在融合机制上,特征编码器一般冻结或者是在端到端的训练中进行微调,回答通常是通过分类器(全连接)或者RNN进行语言的生成。
这种模式也是基于相似分类的方式,训练并寻找CNN图片特征和问题的相关性,通过相关度大小去匹配答案。拥抱transformer,开始尝试多模态表征
VinVL
在视觉语言模型中,视觉端一般都是使用一个预训练的目标检测模型。现在的多模态模型都是聚焦在如何改进跨模态的融合模块上,但是很少聚焦于如何做好目标检测来提升多模态任务。所以,本文聚焦于如何改进以object为中心的视觉表示,用以证明视觉特征在多模态模型中的重要性(尤其是目标检测模型的改进)CLIP
- motivation
先前的网络都是通过训练去预测给定的目标类别(也就是说label是给定的),但是这种监督式的训练会限制网络的泛化能力和可用性(如果出现其他没见过的目标对象还得打上额外的标签)
因此paper提出从关于图片的文本进行学习,以达到利用更多的有监督数据,证明了通过预测哪个caption对应哪张图片的简单预训练任务是学习SOTA图像表征的好方法。这里用到的数据集是从互联网收集的400 million图像文本对数据集。经过预训练后,这些caption的自然语言被reference学习到的视觉概念,能够以零样本的转移学习到下游任务上。 - process
- CLIP 将一批文本通过 Text Encoder 编码成一批 word embedding,将一批图片(与文本一一对应)通过 Image Encoder 编码成一批 feature embedding,然后将对应的 word embedding 和 feature embedding 先归一化然后进行点积得到相似度矩阵,点积数值越大,代表 word embedding 和 feature embedding 的向量越相似,这里的监督信号就是矩阵对角线为 1,其余位置为 0。其中 Text Encoder 使用的是 Transformer,而 Image Encoder 使用 ResNet50 和 ViT 两种架构其中一个,Image Encoder 和 Text Encoder 都是从头训练。
- 然后将预训练好的 CLIP 迁移到下游任务,先将下游任务的标签构建为一批带标签的文本(例如 A photo of a {plane}),然后经过 Text Encoder 编码成一批相应的 word embedding。
- 最后将没有见过的图片进行 zero-shot 预测,通过 Image Encoder 将一张小狗的图片编码成一个 feature embedding,然后跟(2)编码的一批 word embedding 先归一化然后进行点积,最后得到的 logits 中数值最大的位置对应的标签即为最终预测结果。
- 这应该算是一种P-tuning。
- motivation
ViLT
目前参数量最小的多模态Transformer方法。ViLT使用预训练的ViT来初始化交互的transformer,这样就可以直接利用交互层来处理视觉特征,不需要额外增加一个视觉encoder(如Faster-RCNN)。
第一个基于patch projection的多模态预训练模型,其是首个使用patch projection来做visual embedding的方法。
证明了可以将BERT的方法和Vison Transformer结合起来用于多模态transformer。
体现了全词掩码(whole word masking)在预训练时以及图像增强(image augmentations)在微调时的重要性。ALIGN
目前对于多模态的数据集的构建依然严重依赖于昂贵的专家知识。所以,本文作者使用了一个超过10亿的带有噪声的图片文本对,并没有经过数据过滤等进一步处理。
基于对比学习,作者使用了一个非常简单的duel encoder的结构(双塔结构)来学习视觉表示和语言表示。
该模型ALIGN在ImageNet等数据集上取得了非常有竞争力的表现,并且在检索数据集比如Flickr30K和MSCOCO都取得了sota。zero-shot的效果也非常不错。SOHO
用Fast RCNN提取的region特征,是会存在一些问题的:
忽略检测框外的上下文信息;
提取的视觉特定会被局限在目标检测器预定义的类别中;
目标检测器依赖大规模标注数据,并且存在质量低、噪声大等问题。
所以,作者根据这一点,提出了基于grid的预训练模型SOHO。ALBEF
问题一:先有的CLIP和ALIGN等模型虽然通过一些多模态的对比学习等任务,获得了在图像上面性能的大幅度提升,但是并没有学习丰富的多模态交互。问题二:以UNITER为代表的方法使用了多模态encoder来学习联合的图像文本分布。但是他们所学习的文本,图像是没有预先对齐的。
问题三:预训练的数据集大多是由从网络上收集的嘈杂的图像-文本对组成,所以我们现在的预训练目标比如MLM很容易对噪声文本过拟合。
作者通过图片-文本对比学习(ITC)、图片-文本匹配(ITM)、掩码语言模型(MLM),三个预训练任务,并提出动量蒸馏(Momentum Distillation)对抗数据噪音、改进训练过程,在VQA等任务上获得了SOTA。使用了经典的双流模型。image的encoder使用了ViT。Text的encoder比较有意思,并不是用传统的BERT而是只使用了前6层。动量模型 Momentum Model
我也是第一次接触这个词,感觉不是很懂。感觉是使用动量模型给图像-文本对比学习和掩码语言建模生成伪目标。比如给一个图片生成很多描述,然后根据此来进行预训练。比如下图就是给每个图生成了5个伪目标。Probing Inter-modality
作者觉得可以从视觉的方面来改进多模态任务。针对视觉的内部信息的学习以及文本与视觉的多模态学习都被封装在了一个transformer里面,这样是不合理的。所以作者把self-attention引入了视觉端用以促进模态内的学习。(我怎么感觉这个idea,好像在以前哪里就看过,但是咱们不说)接着提出了一个 Inter-Modality Flow (IMF) 的metric用于衡量视觉和语言的融合度。并且提出了一个Visual Directionary(VD)来提取视觉特征。CLIP-ViL
SimVLM
CPT
METER
VLMo
TRAR
MEDVQA的发展
2018年,ImageCLIF发布了视觉问答相关任务,同时提出了数据集ImageCLEF2018 共2866个图片,6413个问答对。次年ImageCLEF2019 共4200个放射性图片,15992个问答对 ,有器官和病理图。
他人总结知乎
元学习时代:Overcoming Data Limitation in Medical Visual Question Answering
提出将元学习用于MEDVQA(MAML(Model-Agnostic Meta-Learning ))
简单来说,就是大哥A和小弟B,A想要做一个2分类,但A和B都不会。于是A先命令B去学习做20分类问题,B学有所成回来后,再找一些2分类来教A。相比起B无依无靠从头学习,对于A来说,B已经是一个好的老师了,所以A学习起来很容易,赢在了起跑线上。用专业的话来说就是A有一个好的初始条件。后续只需要少量特定任务的数据做微调(fine-tune)就能让网络快速收敛增强推理:Medical Visual Question Answering via Conditional Reasoning
基于前文利用元学习在数据和图像处理阶段做出的改进,提出QCR(question-conditional reasoning)和TCR(type-conditional reasoning)模块进一步获取问题中的关键信息。
视觉/语言特征提取,特征融合和预测。第一篇工作通过元学习在视觉特征提取这部分做出了贡献。但医疗视觉问答的困难还包括对于问题的理解。封闭式问题需要的推理较少,更容易理解,而开放式的问题推理较多,更难理解。现有的方法在开放式问题上表现都不好。作者认为这是由于过去的模型用同样的方法理解这两种问题,没有对具体的问题具体分析。并且这些问题里包含的丰富信息并没有被充分利用。
QCR: Question-Conditioned Reasoning Module
QCR是为了从问题句子中学习到有关问题形式(开放/非开放)以及问题种类(VQA-RAD的11类问题,见前文)的信息来帮助特征融合过程中筛选出图像中对应的有效注意力区域,排除无关信息。
TCR: Type-Conditioned Reasoning Module
对于简单的封闭式问题和复杂的开放式问题,文章希望用两个模块分开处理,这样模型再遇到未知问题时,能有更好的多尺度推理能力。
因此TCR模块被用于预先处理问题句子。它的内部结构和QCR很像。在得到词嵌入特征后,用MLP去获得这个特征的一个标量表示(或者说分数)。因为开放式和封闭式问题具有明显的词语却别:封闭式一般以“Is\Dose\Are”开头,而开放式一般以“What\How many\Where”开头。所以MLP能够很好的区分这两种特征。最后用softmax函数得到一个二分类概率。如果概率为1,则网络训练出一个针对开放式问题有效的QCR模块;如果概率为0,则网络训练一个仅针对封闭式问题有效的QCR模块。
- 跨模态自注意力的使用:Cross-Modal Self-Attention with Multi-Task Pre-Training for Medical Visual Question Answering
此文基于集成学习方法,在前文使用的自注意力模型的基础上使用了多任务预训练模型,不是提高模态交互能力而是提高视觉编码的准确率从而提高整体模型的一个性能(MTPT)
三个在不同任务(CT,X-ray,MRI)上预训练的resnet共同对视觉特征进行编码,然后对三个编码向量进行加权相加得到融合编码,源代码: distmodal_emb = [modal_classifier, abd_v_emb, brain_v_emb, chest_v_emb] 其中modal_classifier使用了医学上常用的MCNET
- CLIP预训练模型:Does CLIP Benefit Visual Question Answering in the Medical Domain as Much as it Does in the General Domain?
在2.的基础上修改了图像编码器改成clip,获得了良好的跨模态交互性能,并且基于预训练数据获得了医学图像更好的特征表示。
医学影像上的VQA
视频介绍
与自然图像VQA对比
| 样本区别 | 医学图像 | 自然图像 |
|---|---|---|
| 数量上 | 样本数据量少,无法人工产生 | 可以由人工制作而成 |
| 信息上 | 噪声大,关键信息占比小 | 噪声可控,信息充足 |
| 标注上 | 专业人员标注,难度大 | 普通人员可以标注 |
| 特征上 | 整体相似度高,难以区分,但细节十分多样 | 有明显特征 |
| 结构上 | 多数为灰度图,放射图,光谱单一 | 复杂光谱 |
我的一些观察(领域整理)
有效融合图像文本信息的三种方法
特征拼接concat | 跨模态注意 | 条件批量归一化(CBN)
科普思路
-
多模态信息整合
如同人在不同环境下对物理做定位那样,亮的时候更依赖视觉,暗的时候更依赖听觉,所以在多模态信息整合的过程中,有所谓的信息筛选,加权的形式(计算贝叶斯后验概率),这刚好也是神经网络擅长的。
不同的模态间可以产生一定的互补作用。比如去同一空间的表征均值。同时,对信息的整合也可以提高大脑对估计的可靠程度。分布式多模态信息整合系统
分布式架构中的处理器可位于不同的物理位置,例如分布于大脑中的不同脑区。总体而言,分布式系统有以下几个特点:- 每个处理器可以采用模块化构造,即处理器内部的结构都一致;
- 处理器可以分布于不同的物理位置,但处理器之间需要相互通信;
- 分布式系统中,处理器实现了平行分布式计算,它们同时完成了信息整合;
- 由于没有处理器位于系统拓扑结构的中心,分布式系统鲁棒性(robustness)很强。即失去一个或多个处理器,其余完好的处理器仍然可以进行信息整合工作。试想,计算机程序能不能模拟出这样的网络。
信息分离
多模态信息整合与分离的道理看似简单,但在具体实现这一计算任务时却面临一个根本性的难题,即大脑事先并不知晓这些输入的来源及产生过程,从而不确定是到底应该进行信息整合还是分离。在计算上大脑面临的是一个“鸡生蛋”还是“蛋生鸡”的挑战:如果不整合多模态信息,那么大脑可能无法对外界世界做出准确估计;但如果大脑简单且随意地整合信息,大脑就可能犯错,把不是来自同一物体的信息整合在一起从而得出错误的结论,即张冠李戴。那么大脑的神经环路如何同时进行整合与分离?它们对应的神经基础是什么?在视觉前庭脑区(MSTd区和VIP区),实验发现神经元可以根据它们的调谐曲线分成两类[6][12]。其中一类神经元在两种模态下的刺激偏好一致,被称为一致性神经元(congruent neuron),见图6A。图2C和E所示的理论及实验都已证实一致性神经元负责视觉和前庭输入的多模态信息整合。而出人意料的是,除了一致性神经元之外,多模态脑区中还存在另一类神经元,它在两种模态下的刺激偏好几乎完全相反,被称为反向神经元(opposite neuron),如图6B所示。例如,若一个反向神经元偏好0度的视觉运动方向,那么它会偏好180度的前庭刺激运动方向。
主流方法
- 多模态表示:联合表示是投影到同一个空间,或者用协同表示方法将不同的空间加上关联(相关性约束)
- 模态转化:同样的一个信息,在不同的表达之间转化
- 图文编码转化
- 对齐:找信息之中的局部关联性,给他们自身或者整体加上关系
- Attention
- 多模态融合:数据融合,特征融合,结果融合
- 数据融合:牛
- 特征:torch.cat/torch.sum/torch.mul、注意力、双线性
- 结果:投票,均值方差
- 协同学习(打辅助):使用一个资源丰富的模态信息来辅助另一个资源相对贫瘠的模态进行学习,协同学习是与需要解决的任务无关的,因此它可以用于辅助多模态映射、融合及对齐等问题的研究。
领域综述
- 主要来自2021年的一篇综述《Medical Visual Question Answering: A Survey》
- 因为如上医学的图像的数据特殊性,视觉编码器大家在一开始使用了预训练迁移学习和模型不可知元学习的方法(简单的总结一下MAML的基本思路,即寻找一个优化的参数θ,这个参数对于相关任务是通用的,其能够帮助我们使用更少量的样本进行学习,缩短训练时间)以及采用自编码器进行去噪。但也正因为医学问答中的数据标签少,预训练模型就有了很高的可验证性,这也是一个可以做工作的地方
没有大规模并且具有鲁棒性的数据集是医学问答无法直接应用现有流行VQA方法的一大痛点。更多是采用CNN等结构进行特征提取。
样本上的区别:
- 样本数据量少,并且无法人工生成:医学图像只能来源于病患,并且,无法人工产生。从医学伦理角度,我们不可能人为制造病患来收集医学图像。在其他领域,往往可以人工生成样本数据。这是最大的区别点。
- 噪声大,关键信息占比小:医学图像中,关键信息往往是细节,比如肺癌图片,有无肺癌结节才是关键信息,肺癌结节的亮点在整个肺癌图像上面积占比不到1%、甚至更低,所以,医学图像的信噪比很低。在其他领域的图像,要识别的对象往往是占主体的,所以,其他领域图像的信噪比往往较高。
- 标注难度大 ,成本高:医学图像的标注,必须是医学专业的人员,标注数据极难获得(有时候,这都不是花钱就能解决的问题)。其他领域的图像,往往可以请普通人标注,甚至借助深度学习技术来标注,标注成本低。标注成本高也决定了医学图像样本数据量不可能太大。
- 研究人员少,前沿技术难以应用:能够接触医学图像的图像处理的研究人员范围很小,一般来说,最前沿的图像处理技术是针对其他领域的来设计的,当这些技术应用到医学图像领域时,往往会出现适应性的问题,所以,医学图像处理的技术水平往往也不是最高的。
我的一些前期工作
mmf
fackbook开发的针对多模态任务的集成训练工具,代码封装性高,极其复杂,但可以同时接触到更多的模型和更好的数据集
Bert read code
跟读了一遍bert文本分类代码,但因为没有实际的任务场景,很快又忘记了
Vilt
这里有vit+bert以及将其轻量化的视觉编码器
BAN+MED 阅读
搞懂模型和代码
- 导入数据,得到对应的feature(dataset process)
- 一些基本的初始化参数确定后,模型会调用class VQAFeatureDataset(Dataset)获取特征
- 初始化函数:导入ans和label的索引关系pkl(临时存储的变量关系)
- 定义VQAfeature类的使用方法:tokenize()\tensorize()
- 设计默认类getitem,读取npy返回各类型的数据
- 一些基本的初始化参数确定后,模型会调用class VQAFeatureDataset(Dataset)获取特征
- 模型设计
- BAN模型(QCR-TCR)
- 初始化:w_emb、q_emb、att 建立模型时直接获得dateset中的数据编码形式,采用的是golve编码,再将dataset中的v_dim数据以及给定的h_dim初始化注意力和BiRESNET还有FC
- 根据参数选择DAE、MAML两种方式调整编码的向量形式,以及是否做cat
- 内部结构
- Embedding层(W,Q)
- 针对封闭域问答的分类层:BiAttention + BiResNet + SimpleClassifier
点击此处打开折叠代码
- 针对开放域问答的分类层:BiAttention + BiResNet + SimpleClassifier1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
69
70
71
72
73
74
75
76
77
78
79
80
81(close_att): BiAttention(
(logits): BCNet(
(v_net): FCNet(
(main): Sequential(
(0): Dropout(p=0.2, inplace=False)
(1): Linear(in_features=128, out_features=3072, bias=True)
(2): ReLU()
)
)
(q_net): FCNet(
(main): Sequential(
(0): Dropout(p=0.2, inplace=False)
(1): Linear(in_features=1024, out_features=3072, bias=True)
(2): ReLU()
)
)
(dropout): Dropout(p=0.5, inplace=False)
(p_net): AvgPool1d(kernel_size=(3,), stride=(3,), padding=(0,))
)
)
(close_resnet): BiResNet(
(b_net): ModuleList(
(0): BCNet(
(v_net): FCNet(
(main): Sequential(
(0): Dropout(p=0.2, inplace=False)
(1): Linear(in_features=128, out_features=1024, bias=True)
(2): ReLU()
)
)
(q_net): FCNet(
(main): Sequential(
(0): Dropout(p=0.2, inplace=False)
(1): Linear(in_features=1024, out_features=1024, bias=True)
(2): ReLU()
)
)
(dropout): Dropout(p=0.5, inplace=False)
)
(1): BCNet(
(v_net): FCNet(
(main): Sequential(
(0): Dropout(p=0.2, inplace=False)
(1): Linear(in_features=128, out_features=1024, bias=True)
(2): ReLU()
)
)
(q_net): FCNet(
(main): Sequential(
(0): Dropout(p=0.2, inplace=False)
(1): Linear(in_features=1024, out_features=1024, bias=True)
(2): ReLU()
)
)
(dropout): Dropout(p=0.5, inplace=False)
)
)
(q_prj): ModuleList(
(0): FCNet(
(main): Sequential(
(0): Dropout(p=0.2, inplace=False)
(1): Linear(in_features=1024, out_features=1024, bias=True)
)
)
(1): FCNet(
(main): Sequential(
(0): Dropout(p=0.2, inplace=False)
(1): Linear(in_features=1024, out_features=1024, bias=True)
)
)
)
(c_prj): ModuleList()
)
(close_classifier): SimpleClassifier(
(main): Sequential(
(0): Linear(in_features=1024, out_features=2048, bias=True)
(1): ReLU()
(2): Dropout(p=0.5, inplace=True)
(3): Linear(in_features=2048, out_features=56, bias=True)
)
)- 模型交互层typeatt1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
69
70
71
72
73
74
75
76
77
78
79
80
81(open_att): BiAttention(
(logits): BCNet(
(v_net): FCNet(
(main): Sequential(
(0): Dropout(p=0.2, inplace=False)
(1): Linear(in_features=128, out_features=3072, bias=True)
(2): ReLU()
)
)
(q_net): FCNet(
(main): Sequential(
(0): Dropout(p=0.2, inplace=False)
(1): Linear(in_features=1024, out_features=3072, bias=True)
(2): ReLU()
)
)
(dropout): Dropout(p=0.5, inplace=False)
(p_net): AvgPool1d(kernel_size=(3,), stride=(3,), padding=(0,))
)
)
(open_resnet): BiResNet(
(b_net): ModuleList(
(0): BCNet(
(v_net): FCNet(
(main): Sequential(
(0): Dropout(p=0.2, inplace=False)
(1): Linear(in_features=128, out_features=1024, bias=True)
(2): ReLU()
)
)
(q_net): FCNet(
(main): Sequential(
(0): Dropout(p=0.2, inplace=False)
(1): Linear(in_features=1024, out_features=1024, bias=True)
(2): ReLU()
)
)
(dropout): Dropout(p=0.5, inplace=False)
)
(1): BCNet(
(v_net): FCNet(
(main): Sequential(
(0): Dropout(p=0.2, inplace=False)
(1): Linear(in_features=128, out_features=1024, bias=True)
(2): ReLU()
)
)
(q_net): FCNet(
(main): Sequential(
(0): Dropout(p=0.2, inplace=False)
(1): Linear(in_features=1024, out_features=1024, bias=True)
(2): ReLU()
)
)
(dropout): Dropout(p=0.5, inplace=False)
)
)
(q_prj): ModuleList(
(0): FCNet(
(main): Sequential(
(0): Dropout(p=0.2, inplace=False)
(1): Linear(in_features=1024, out_features=1024, bias=True)
)
)
(1): FCNet(
(main): Sequential(
(0): Dropout(p=0.2, inplace=False)
(1): Linear(in_features=1024, out_features=1024, bias=True)
)
)
)
(c_prj): ModuleList()
)
(open_classifier): SimpleClassifier(
(main): Sequential(
(0): Linear(in_features=1024, out_features=2048, bias=True)
(1): ReLU()
(2): Dropout(p=0.5, inplace=True)
(3): Linear(in_features=2048, out_features=431, bias=True)
)
)- 元学习编码MAML1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17(typeatt): typeAttention(
(w_emb): WordEmbedding(
(emb): Embedding(1178, 300, padding_idx=1177)
(dropout): Dropout(p=0.0, inplace=False)
)
(q_emb): QuestionEmbedding(
(rnn): GRU(300, 1024, batch_first=True)
)
(q_final): QuestionAttention(
(tanh_gate): Linear(in_features=1324, out_features=1024, bias=True)
(sigmoid_gate): Linear(in_features=1324, out_features=1024, bias=True)
(attn): Linear(in_features=1024, out_features=1, bias=True)
)
(f_fc1): Linear(in_features=1024, out_features=2048, bias=True)
(f_fc2): Linear(in_features=2048, out_features=1024, bias=True)
(f_fc3): Linear(in_features=1024, out_features=1024, bias=True)
)- 自编码器DAE1
2
3
4
5
6
7
8
9
10(maml): SimpleCNN(
(conv1): Conv2d(1, 64, kernel_size=(3, 3), stride=(2, 2), padding=(1, 1))
(conv1_bn): BatchNorm2d(64, eps=1e-05, momentum=0.05, affine=True, track_running_stats=True)
(conv2): Conv2d(64, 64, kernel_size=(3, 3), stride=(2, 2), padding=(1, 1))
(conv2_bn): BatchNorm2d(64, eps=1e-05, momentum=0.05, affine=True, track_running_stats=True)
(conv3): Conv2d(64, 64, kernel_size=(3, 3), stride=(2, 2), padding=(1, 1))
(conv3_bn): BatchNorm2d(64, eps=1e-05, momentum=0.05, affine=True, track_running_stats=True)
(conv4): Conv2d(64, 64, kernel_size=(3, 3), stride=(2, 2), padding=(1, 1))
(conv4_bn): BatchNorm2d(64, eps=1e-05, momentum=0.05, affine=True, track_running_stats=True)
)1
2
3
4
5
6
7
8
9
10
11
12(ae): Auto_Encoder_Model(
(conv1): Conv2d(1, 64, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))
(max_pool1): MaxPool2d(kernel_size=2, stride=2, padding=0, dilation=1, ceil_mode=False)
(conv2): Conv2d(64, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))
(max_pool2): MaxPool2d(kernel_size=2, stride=2, padding=0, dilation=1, ceil_mode=False)
(conv3): Conv2d(32, 16, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))
(tran_conv1): ConvTranspose2d(16, 32, kernel_size=(3, 3), stride=(2, 2), padding=(1, 1), output_padding=(1, 1))
(conv4): Conv2d(32, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))
(tran_conv2): ConvTranspose2d(32, 64, kernel_size=(3, 3), stride=(2, 2), padding=(1, 1), output_padding=(1, 1))
(conv5): Conv2d(64, 1, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))
)
(convert): Linear(in_features=16384, out_features=64, bias=True)
- BAN模型(QCR-TCR)
- CCMSA模型
- 模型结构
<details>
<summary>点击此处打开折叠代码</summary>
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
69
70
71
72
73
74
75
76
77
78
79
80
81
82
83
84
85
86
87
88
89
90
91
92
93
94
95
96
97
98
99
100
101
102
103
104
105
106
107
108
109
110
111
112
113
114
115
116
117
118
119
120
121
122
123
124
125
126
127
128
129
130
131
132
133
134
135
136
137
138
139
140
141
142
143
144
145
146
147
148
149
150
151
152
153
154
155
156
157
158
159
160
161
162
163
164
165
166
167
168
169
170
171
172
173
174
175
176
177
178
179
180
181
182
183
184
185
186
187
188
189
190
191
192
193
194
195
196
197
198
199
200
201
202
203
204
205
206
207
208
209
210
211
212
213
214
215
216
217
218
219
220
221
222
223
224
225
226
227
228
229
230
231
232
233
234
235
236
237
238
239
240
241
242
243
244
245
246
247
248
249
250
251
252
253
254
255
256
257
258
259
260
261
262
263
264
265
266
267
268
269
270
271
272
273
274
275
276
277
278
279
280
281
282
283
284
285
286
287
288
289
290
291
292
293
294
295
296
297
298
299
300
301
302
303
304
305
306
307
308
309
310
311
312
313
314
315
316
317
318
319
320
321
322
323
324
325
326
327
328
329
330
331
332
333
334
335
336
337
338
339
340
341
342
343
344
345
346
347
348
349
350
351
352
353
354
355
356
357
358
359
360
361
362
363
364
365
366
367
368
369
370
371
372
373
374
375
376
377
378
379
380
381
382
383
384
385
386
387
388
389
390
391
392
393
394
395
396
397
398
399
400
401
402
403
404
405
406
407
408
409
410
411
412
CCMSA_Model(
(w_emb): WordEmbedding(
(emb): Embedding(1178, 300, padding_idx=1177)
(emb_): Embedding(1178, 300, padding_idx=1177)
(dropout): Dropout(p=0.0, inplace=False)
)
(q_emb): QuestionEmbedding(
(rnn): LSTM(600, 1024, batch_first=True)
)
(cmsa0): NONLocalBlock3D(
(g): Conv3d(2184, 1092, kernel_size=(1, 1, 1), stride=(1, 1, 1))
(W): Sequential(
(0): Conv3d(1092, 2184, kernel_size=(1, 1, 1), stride=(1, 1, 1))
(1): BatchNorm3d(2184, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
)
(theta): Conv3d(2184, 1092, kernel_size=(1, 1, 1), stride=(1, 1, 1))
(phi): Conv3d(2184, 1092, kernel_size=(1, 1, 1), stride=(1, 1, 1))
)
(cmsa1): NONLocalBlock3D(
(g): Conv3d(2184, 1092, kernel_size=(1, 1, 1), stride=(1, 1, 1))
(W): Sequential(
(0): Conv3d(1092, 2184, kernel_size=(1, 1, 1), stride=(1, 1, 1))
(1): BatchNorm3d(2184, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
)
(theta): Conv3d(2184, 1092, kernel_size=(1, 1, 1), stride=(1, 1, 1))
(phi): Conv3d(2184, 1092, kernel_size=(1, 1, 1), stride=(1, 1, 1))
)
(fc): Linear(in_features=2184, out_features=1024, bias=True)
(classifier): BayesClassifier(
(fc1): BayesLinear_Normalq()
(fc2): BayesLinear_Normalq()
(activate): ReLU()
)
(maml_v_emb): SimpleCNN(
(conv1): Conv2d(1, 64, kernel_size=(3, 3), stride=(2, 2), padding=(1, 1))
(conv1_bn): BatchNorm2d(64, eps=1e-05, momentum=0.05, affine=True, track_running_stats=True)
(conv2): Conv2d(64, 64, kernel_size=(3, 3), stride=(2, 2), padding=(1, 1))
(conv2_bn): BatchNorm2d(64, eps=1e-05, momentum=0.05, affine=True, track_running_stats=True)
(conv3): Conv2d(64, 64, kernel_size=(3, 3), stride=(2, 2), padding=(1, 1))
(conv3_bn): BatchNorm2d(64, eps=1e-05, momentum=0.05, affine=True, track_running_stats=True)
(conv4): Conv2d(64, 64, kernel_size=(3, 3), stride=(2, 2), padding=(1, 1))
(conv4_bn): BatchNorm2d(64, eps=1e-05, momentum=0.05, affine=True, track_running_stats=True)
)
(ae_v_emb): Auto_Encoder_Model(
(conv1): Conv2d(1, 64, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))
(max_pool1): MaxPool2d(kernel_size=2, stride=2, padding=0, dilation=1, ceil_mode=False)
(conv2): Conv2d(64, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))
(max_pool2): MaxPool2d(kernel_size=2, stride=2, padding=0, dilation=1, ceil_mode=False)
(conv3): Conv2d(32, 16, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))
(tran_conv1): ConvTranspose2d(16, 32, kernel_size=(3, 3), stride=(2, 2), padding=(1, 1), output_padding=(1, 1))
(conv4): Conv2d(32, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))
(tran_conv2): ConvTranspose2d(32, 64, kernel_size=(3, 3), stride=(2, 2), padding=(1, 1), output_padding=(1, 1))
(conv5): Conv2d(64, 1, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))
)
(convert): Linear(in_features=16384, out_features=64, bias=True)
(clip_v_emb): CLIP(
(visual): ModifiedResNet(
(conv1): Conv2d(3, 32, kernel_size=(3, 3), stride=(2, 2), padding=(1, 1), bias=False)
(bn1): BatchNorm2d(32, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(conv2): Conv2d(32, 32, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): BatchNorm2d(32, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(conv3): Conv2d(32, 64, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn3): BatchNorm2d(64, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(avgpool): AvgPool2d(kernel_size=2, stride=2, padding=0)
(relu): ReLU(inplace=True)
(layer1): Sequential(
(0): Bottleneck(
(conv1): Conv2d(64, 64, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): BatchNorm2d(64, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(conv2): Conv2d(64, 64, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): BatchNorm2d(64, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(avgpool): Identity()
(conv3): Conv2d(64, 256, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): BatchNorm2d(256, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu): ReLU(inplace=True)
(downsample): Sequential(
(-1): AvgPool2d(kernel_size=1, stride=1, padding=0)
(0): Conv2d(64, 256, kernel_size=(1, 1), stride=(1, 1), bias=False)
(1): BatchNorm2d(256, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
)
)
(1): Bottleneck(
(conv1): Conv2d(256, 64, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): BatchNorm2d(64, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(conv2): Conv2d(64, 64, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): BatchNorm2d(64, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(avgpool): Identity()
(conv3): Conv2d(64, 256, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): BatchNorm2d(256, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu): ReLU(inplace=True)
)
(2): Bottleneck(
(conv1): Conv2d(256, 64, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): BatchNorm2d(64, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(conv2): Conv2d(64, 64, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): BatchNorm2d(64, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(avgpool): Identity()
(conv3): Conv2d(64, 256, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): BatchNorm2d(256, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu): ReLU(inplace=True)
)
)
(layer2): Sequential(
(0): Bottleneck(
(conv1): Conv2d(256, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(conv2): Conv2d(128, 128, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(avgpool): AvgPool2d(kernel_size=2, stride=2, padding=0)
(conv3): Conv2d(128, 512, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): BatchNorm2d(512, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu): ReLU(inplace=True)
(downsample): Sequential(
(-1): AvgPool2d(kernel_size=2, stride=2, padding=0)
(0): Conv2d(256, 512, kernel_size=(1, 1), stride=(1, 1), bias=False)
(1): BatchNorm2d(512, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
)
)
(1): Bottleneck(
(conv1): Conv2d(512, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(conv2): Conv2d(128, 128, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(avgpool): Identity()
(conv3): Conv2d(128, 512, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): BatchNorm2d(512, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu): ReLU(inplace=True)
)
(2): Bottleneck(
(conv1): Conv2d(512, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(conv2): Conv2d(128, 128, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(avgpool): Identity()
(conv3): Conv2d(128, 512, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): BatchNorm2d(512, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu): ReLU(inplace=True)
)
(3): Bottleneck(
(conv1): Conv2d(512, 128, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(conv2): Conv2d(128, 128, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(avgpool): Identity()
(conv3): Conv2d(128, 512, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): BatchNorm2d(512, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu): ReLU(inplace=True)
)
)
(layer3): Sequential(
(0): Bottleneck(
(conv1): Conv2d(512, 256, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): BatchNorm2d(256, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(conv2): Conv2d(256, 256, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): BatchNorm2d(256, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(avgpool): AvgPool2d(kernel_size=2, stride=2, padding=0)
(conv3): Conv2d(256, 1024, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): BatchNorm2d(1024, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu): ReLU(inplace=True)
(downsample): Sequential(
(-1): AvgPool2d(kernel_size=2, stride=2, padding=0)
(0): Conv2d(512, 1024, kernel_size=(1, 1), stride=(1, 1), bias=False)
(1): BatchNorm2d(1024, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
)
)
(1): Bottleneck(
(conv1): Conv2d(1024, 256, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): BatchNorm2d(256, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(conv2): Conv2d(256, 256, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): BatchNorm2d(256, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(avgpool): Identity()
(conv3): Conv2d(256, 1024, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): BatchNorm2d(1024, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu): ReLU(inplace=True)
)
(2): Bottleneck(
(conv1): Conv2d(1024, 256, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): BatchNorm2d(256, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(conv2): Conv2d(256, 256, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): BatchNorm2d(256, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(avgpool): Identity()
(conv3): Conv2d(256, 1024, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): BatchNorm2d(1024, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu): ReLU(inplace=True)
)
(3): Bottleneck(
(conv1): Conv2d(1024, 256, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): BatchNorm2d(256, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(conv2): Conv2d(256, 256, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): BatchNorm2d(256, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(avgpool): Identity()
(conv3): Conv2d(256, 1024, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): BatchNorm2d(1024, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu): ReLU(inplace=True)
)
(4): Bottleneck(
(conv1): Conv2d(1024, 256, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): BatchNorm2d(256, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(conv2): Conv2d(256, 256, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): BatchNorm2d(256, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(avgpool): Identity()
(conv3): Conv2d(256, 1024, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): BatchNorm2d(1024, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu): ReLU(inplace=True)
)
(5): Bottleneck(
(conv1): Conv2d(1024, 256, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): BatchNorm2d(256, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(conv2): Conv2d(256, 256, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): BatchNorm2d(256, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(avgpool): Identity()
(conv3): Conv2d(256, 1024, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): BatchNorm2d(1024, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu): ReLU(inplace=True)
)
)
(layer4): Sequential(
(0): Bottleneck(
(conv1): Conv2d(1024, 512, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): BatchNorm2d(512, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(conv2): Conv2d(512, 512, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): BatchNorm2d(512, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(avgpool): AvgPool2d(kernel_size=2, stride=2, padding=0)
(conv3): Conv2d(512, 2048, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): BatchNorm2d(2048, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu): ReLU(inplace=True)
(downsample): Sequential(
(-1): AvgPool2d(kernel_size=2, stride=2, padding=0)
(0): Conv2d(1024, 2048, kernel_size=(1, 1), stride=(1, 1), bias=False)
(1): BatchNorm2d(2048, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
)
)
(1): Bottleneck(
(conv1): Conv2d(2048, 512, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): BatchNorm2d(512, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(conv2): Conv2d(512, 512, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): BatchNorm2d(512, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(avgpool): Identity()
(conv3): Conv2d(512, 2048, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): BatchNorm2d(2048, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu): ReLU(inplace=True)
)
(2): Bottleneck(
(conv1): Conv2d(2048, 512, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn1): BatchNorm2d(512, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(conv2): Conv2d(512, 512, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1), bias=False)
(bn2): BatchNorm2d(512, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(avgpool): Identity()
(conv3): Conv2d(512, 2048, kernel_size=(1, 1), stride=(1, 1), bias=False)
(bn3): BatchNorm2d(2048, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)
(relu): ReLU(inplace=True)
)
)
(attnpool): AttentionPool2d(
(k_proj): Linear(in_features=2048, out_features=2048, bias=True)
(q_proj): Linear(in_features=2048, out_features=2048, bias=True)
(v_proj): Linear(in_features=2048, out_features=2048, bias=True)
(c_proj): Linear(in_features=2048, out_features=1024, bias=True)
)
)
(transformer): Transformer(
(resblocks): Sequential(
(0): ResidualAttentionBlock(
(attn): MultiheadAttention(
(out_proj): _LinearWithBias(in_features=512, out_features=512, bias=True)
)
(ln_1): LayerNorm((512,), eps=1e-05, elementwise_affine=True)
(mlp): Sequential(
(c_fc): Linear(in_features=512, out_features=2048, bias=True)
(gelu): QuickGELU()
(c_proj): Linear(in_features=2048, out_features=512, bias=True)
)
(ln_2): LayerNorm((512,), eps=1e-05, elementwise_affine=True)
)
(1): ResidualAttentionBlock(
(attn): MultiheadAttention(
(out_proj): _LinearWithBias(in_features=512, out_features=512, bias=True)
)
(ln_1): LayerNorm((512,), eps=1e-05, elementwise_affine=True)
(mlp): Sequential(
(c_fc): Linear(in_features=512, out_features=2048, bias=True)
(gelu): QuickGELU()
(c_proj): Linear(in_features=2048, out_features=512, bias=True)
)
(ln_2): LayerNorm((512,), eps=1e-05, elementwise_affine=True)
)
(2): ResidualAttentionBlock(
(attn): MultiheadAttention(
(out_proj): _LinearWithBias(in_features=512, out_features=512, bias=True)
)
(ln_1): LayerNorm((512,), eps=1e-05, elementwise_affine=True)
(mlp): Sequential(
(c_fc): Linear(in_features=512, out_features=2048, bias=True)
(gelu): QuickGELU()
(c_proj): Linear(in_features=2048, out_features=512, bias=True)
)
(ln_2): LayerNorm((512,), eps=1e-05, elementwise_affine=True)
)
(3): ResidualAttentionBlock(
(attn): MultiheadAttention(
(out_proj): _LinearWithBias(in_features=512, out_features=512, bias=True)
)
(ln_1): LayerNorm((512,), eps=1e-05, elementwise_affine=True)
(mlp): Sequential(
(c_fc): Linear(in_features=512, out_features=2048, bias=True)
(gelu): QuickGELU()
(c_proj): Linear(in_features=2048, out_features=512, bias=True)
)
(ln_2): LayerNorm((512,), eps=1e-05, elementwise_affine=True)
)
(4): ResidualAttentionBlock(
(attn): MultiheadAttention(
(out_proj): _LinearWithBias(in_features=512, out_features=512, bias=True)
)
(ln_1): LayerNorm((512,), eps=1e-05, elementwise_affine=True)
(mlp): Sequential(
(c_fc): Linear(in_features=512, out_features=2048, bias=True)
(gelu): QuickGELU()
(c_proj): Linear(in_features=2048, out_features=512, bias=True)
)
(ln_2): LayerNorm((512,), eps=1e-05, elementwise_affine=True)
)
(5): ResidualAttentionBlock(
(attn): MultiheadAttention(
(out_proj): _LinearWithBias(in_features=512, out_features=512, bias=True)
)
(ln_1): LayerNorm((512,), eps=1e-05, elementwise_affine=True)
(mlp): Sequential(
(c_fc): Linear(in_features=512, out_features=2048, bias=True)
(gelu): QuickGELU()
(c_proj): Linear(in_features=2048, out_features=512, bias=True)
)
(ln_2): LayerNorm((512,), eps=1e-05, elementwise_affine=True)
)
(6): ResidualAttentionBlock(
(attn): MultiheadAttention(
(out_proj): _LinearWithBias(in_features=512, out_features=512, bias=True)
)
(ln_1): LayerNorm((512,), eps=1e-05, elementwise_affine=True)
(mlp): Sequential(
(c_fc): Linear(in_features=512, out_features=2048, bias=True)
(gelu): QuickGELU()
(c_proj): Linear(in_features=2048, out_features=512, bias=True)
)
(ln_2): LayerNorm((512,), eps=1e-05, elementwise_affine=True)
)
(7): ResidualAttentionBlock(
(attn): MultiheadAttention(
(out_proj): _LinearWithBias(in_features=512, out_features=512, bias=True)
)
(ln_1): LayerNorm((512,), eps=1e-05, elementwise_affine=True)
(mlp): Sequential(
(c_fc): Linear(in_features=512, out_features=2048, bias=True)
(gelu): QuickGELU()
(c_proj): Linear(in_features=2048, out_features=512, bias=True)
)
(ln_2): LayerNorm((512,), eps=1e-05, elementwise_affine=True)
)
(8): ResidualAttentionBlock(
(attn): MultiheadAttention(
(out_proj): _LinearWithBias(in_features=512, out_features=512, bias=True)
)
(ln_1): LayerNorm((512,), eps=1e-05, elementwise_affine=True)
(mlp): Sequential(
(c_fc): Linear(in_features=512, out_features=2048, bias=True)
(gelu): QuickGELU()
(c_proj): Linear(in_features=2048, out_features=512, bias=True)
)
(ln_2): LayerNorm((512,), eps=1e-05, elementwise_affine=True)
)
(9): ResidualAttentionBlock(
(attn): MultiheadAttention(
(out_proj): _LinearWithBias(in_features=512, out_features=512, bias=True)
)
(ln_1): LayerNorm((512,), eps=1e-05, elementwise_affine=True)
(mlp): Sequential(
(c_fc): Linear(in_features=512, out_features=2048, bias=True)
(gelu): QuickGELU()
(c_proj): Linear(in_features=2048, out_features=512, bias=True)
)
(ln_2): LayerNorm((512,), eps=1e-05, elementwise_affine=True)
)
(10): ResidualAttentionBlock(
(attn): MultiheadAttention(
(out_proj): _LinearWithBias(in_features=512, out_features=512, bias=True)
)
(ln_1): LayerNorm((512,), eps=1e-05, elementwise_affine=True)
(mlp): Sequential(
(c_fc): Linear(in_features=512, out_features=2048, bias=True)
(gelu): QuickGELU()
(c_proj): Linear(in_features=2048, out_features=512, bias=True)
)
(ln_2): LayerNorm((512,), eps=1e-05, elementwise_affine=True)
)
(11): ResidualAttentionBlock(
(attn): MultiheadAttention(
(out_proj): _LinearWithBias(in_features=512, out_features=512, bias=True)
)
(ln_1): LayerNorm((512,), eps=1e-05, elementwise_affine=True)
(mlp): Sequential(
(c_fc): Linear(in_features=512, out_features=2048, bias=True)
(gelu): QuickGELU()
(c_proj): Linear(in_features=2048, out_features=512, bias=True)
)
(ln_2): LayerNorm((512,), eps=1e-05, elementwise_affine=True)
)
)
)
(token_embedding): Embedding(49408, 512)
(ln_final): LayerNorm((512,), eps=1e-05, elementwise_affine=True)
)
)
</details>
相关数据集
自然领域:
- DAQUAR
- VQA1.0/2.0
- COCOQA
- CLEVR
医学领域:(主要是针对CT,X光,以及核磁共振光谱图)
- VQA-Med(噪音很多,本次不实验)
- CT:
- X-ray:
- MAR:
- SLAKE(VQA-Med Plus)
数据大小:642张医学图像,14K的问答对,5个身体部位类别:头,胸,腹,脖子,盆腔;10类问题:器官,位置,知识,异常,模态,平面,质量,颜色,尺寸,形状。 - VQA-RAD
- VQA-PATH (病理数据集,可以后期补做)
现有方法总结
视觉编码器
- CLIP医学预训练模型:PUBCLIP
- MTPT多任务混合预训练:
- MAML元学习编码器:解决以不同器官异常状态为类别的k-shot n-way分类问题
- 常用降噪模块DAE:用于医学图像降噪
MAML又叫模型不可知元学习,是一种针对小样本领域非常行之有效的方法,在元学习中,我们从大量的相关学习任务中获取一小部分的样本点,然后通过元学习器来生成一个快速的学习器,再通过少量的样本作用在新的相关的任务之上。也就是通过相似任务获得一个较优的网络初始化权重,再进行学习,并且可以极大地减小梯度下降次数,提高学习效率(类比获得基本认知,再精细认知)
算法流程
如何训练初始化权重?
假设模型是f,可以用参数θ描述,找来k个与目标近似的任务Tk,并把它们作为一个batch,对每个任务定义loss,并且通过梯度下降最小化得到θimin,这个就是针对任务Ti的初始化参数。k个都训练后,就可以获得响应的参数集合,在采样下一个batch前,使用元更新或者元优化。并通过梯度下降计算出了相对最优参数,并且通过任务Ti中的参数对应的梯度,更新初始化的随机参数。
这一步可以使得模型的初始化移动到了一个相对最优的位置。这一步就是元步,元更新,元训练。
文本编码器
- RNN
- Bert
- QCR\TCR
模型交互思路
- BiATT
- SANATT
- CMSA注意力混合
主要使用的方法 - typeatt附加模块
待使用的方法 - 跨模态的注意力
实验模型:CSMA-MTPT
基于注意力机制的模态融合方法,其中使用到的方法有自注意力机制,多头注意力机制,注意力机制下的模态融合本质上就是优化特征的权重筛选,强调相关性的特征占比。
实验模型:
可以做一个不同的视觉或者语言编码器组合的影响实验
CLIP-AE-BAN/SAN
CMSA-MTPT/BAN/SAN
MAML-AE-BAN/SAN
多任务损失L端到端的训练医学VQA:
$$ L=L_{\text {spe }}+L_{\text {com }} $$
图像编码:从关注视觉特征表达上入手,提高模型性能
我们使用三个独立的ResNet-34网络在相应的外部数据集上预训练,以分别捕获MRI、CT、X-射线的视觉特征。然后使用一个分类器确定医学图像的种类,并以soft方式选择相应的视觉特征:
$$ v=w_{1} v_{a}+w_{2} v_{h}+w_{3} v_{c} $$
v表示最终视觉特征,va,vh,vc分别表示从解码器的对应腹部、头部和胸部图像的输出特征。w是图像种类分类器的输出向量,表示每个医学图像种类的权重。
此外,为了更好理解和回答有关局部图像定位的问题,我们按照[27]获得有同样分辨率的8维空间特征图t作为视觉特征w。空间特征图t中每个位置的空间向量编码归一化坐标(左上、中间、右下、网格的宽和高)。
文字编码
按照前面的工作[17],每个单词都表示为来自VQA-RAD的一个200维BioWordVec词嵌入和另一个200维增强嵌入的连接。BioWordVec是一种基于PubMed和MeSH的预训练的生物医学词嵌入。每个400维嵌入向量送入LSTM获取问题嵌入q∈ R 12×1024。
运用最新的bert获得更加优质的编码能力不失为一项选择,代码能力上也同时要求更高的能力。
跨模态自注意力
跨模态的注意力方式,多注意力机制,先了解程序运作机制,在从实验中获得改进的方法
我的思考:
特征的本质是一种整合或者抽象化后的信息,可以由向量表示,横看成岭侧成峰,但描述的依旧是这一座有特色的山,不同的视角(维度)的信息可以更好地帮助我们认知这个事务,重构还原其本来的样貌,以及更加深入地了解其本质(从投影挖掘并重建存在于高维度的信息)我认为起机理和一些思考大抵如下:
- 维度冗余现象:cat一组特征向量确实可以得到更优秀的特征表达,但如果是相似的观察或者描述方法依然存在维度同质化的问题,其表现其实就是在该特征的真实描述空间上存在线性相关的基向量。cat方法不会把这两组向量合并,而是作为两组独立的向量存在于空间当中,这样的方式会增加其编码的稀疏特性,降低学习效率以及会影响到模型的训练收敛和最后的拟合结果。要解决这个问题,根据论文巴拉巴拉(其实是我)传统的思路是在同一套编码规则下通过相似性判断和线性约束解决。我的方法是直接把编码特征拍平,由自注意力让网络去选择和表征(也就是学习)其有效表达
- 贝叶斯的奥秘:贝叶斯原理直击认知和学习的本质,没有先验的认知和似然作为联系桥梁后验是不存在的。同时也揭示着解决信息不确定性的通路,就是以一个先验知识作为基础,不断通过证据消除不确定性。
贝叶斯神经网络(BNN)原理
理由
模型在推理层/线性层使用了贝叶斯神经网络模型(BNN),贝叶斯神经网络与传统的点估计网络不同,点估计网络的权重w是一个固定的参数,而贝叶斯神经网络的参数是一个概率分布,从而在权重上引入了不确定性。基于先验再通过贝叶斯反向传播算法得道这些权值的一个后验分布形式。有多种优势XXX,在本问题中,贝叶斯推理方法的加入可以给医学诊断这种具有高风险性,高安全需求的行为带来更多,更充分的实施依据。缓解因为模型推理过程中由于不确定性带来的偏差。以及通过后验分布,可以清晰的得到模型对推理的导向,从而提高了模型推理的可解释性。
数学概念和解释
通过不断的采样预测,本质上也是一种集成预测的方法。而且贝叶斯方法比最大似然估计更适合对小样本数据进行建模,贝叶斯方法中,参数的后验分布通过先验和似然的乘积获得,因此贝叶斯可以在模型中引入先验知识,在样本数据较少的情况下提高了模型的收敛能力,降低小样本训练过拟合的风险。
在贝叶斯反向传播中,由于权值的真实分布往往非常复杂,故采用变分近似的方法来对权值的后验分布进行近似估计。就是为每个权值设定一个变分后验,然后通过最小化变分后验和真实后验之间的KL散度从而达到近似的效果。便于神经网络进行训练。
分偶然不确定性和感知不确定性,偶然不确定性存在于数据产生所自带偏差中,是固有的,在模型训练时就会将其作为知识学习进去。感知不确定性是预测时产生的,只与模型相关,可能是因为学习不充分所导致,理论上可以被消除。
代码通读以及实验
构建模型中必须注意的维度匹配问题
多模态模型中,往往都是定义好了所有的channal和维度要求,再喂响应的数据进行学习
所以,视觉编码器的维度定义在args里,有多种,也有混合,模型会根据这个参数来定义一些编码器,注意力层,卷积层,全连接的维度。编码层就按照这个维度去编码得到这个维度的向量,然后给后续层进行处理,千万注意对应关系。
文字编码器同理,LSTM的in,hid,out_dim都对齐文字的编码维度,比如glove,bio等编码,然后和注意力层相连接。
样例:我在代码中常犯的一些问题,v_emb和q_emb分别与FCnet的对齐关系(FCnet在定义时就有indim,outdim)所以我的v,q输入维度必须和这几个参数对齐
解读代码和其中的数学原理
数据处理部分(序列化,还有获取标签返回)
- 这部分就是dataset_RAD.py中的一系列class和处理函数,针对的是一些处理过的数据集,例如将图片和问答对序列化,然后_load_data,
- 图片中根据自己所用的方法去选用不用的裁切方式,例如采用元学习时,将图片进行最小裁切成8484或者128128
- 在定义时还要给图片的维数做一个定义,好拼接后续模型
- 获得标签/匹配对(getitem)
模型搭建部分(图像编码器,语言编码器)
- 图像编码器中可以使用预训练好的编码模型,例如独立的CLIP,maml,DAE以及CMSA论文的三集成MTPT编码器,等方式
- 语言编码器可以用LSTM和纯注意力,或者使用Bert(自用)
特征融合部分
- 采用注意力融合的方式
- 采用QCR-TCR的分段汇总方式
推理部分
- 两层全连接,经典分类器
- 贝叶斯全连接层(把输出的分布都推理出来)
训练部分
- 多个loss的合计会带来什么问题
验证部分
实验流程
可控制变量(实验)一览:
- 选用不同的视觉编码器以及其组合
- Maml
- DAE
- CLIP
- VIT32(论文中在SLAKE上表现最优)
- RN50(论文中在RAD上表现最优)
- RN50*4
- 选用不用的文字编码器
- LSTM
- Self Attention
- 选用不同的模态交互注意力
- SAN
- BAN
- CMSA
- 选用不同的分类器求loss
- FC
- Bayes-FC
- 贝叶斯先验分布(prior.py)形式:球形高斯分布(sigma方差,设定值为0.1)
- loss占比alpha(默认设定值为1)
- 额外
- loss控制是否采用dwa算法
- 是否加入多头注意力层
- 对问题采不采用QCR-TCR(有点麻烦)
- 选用不同的视觉编码器以及其组合
设计实验
实验一:通过准确率说明我的注意力模型构建方法(MEVF-CMSA-plus版本)在融合不同视觉编码器上更有效,缓解模态信息融合中存在的信息干扰问题(信息强化公式)
实验思路:单个编码器下的性能实验(MAMLDAE-BAN | MAMLDAE-CMSA),多个编码器性能实验(MAMLDAECLIP-BAN | MAMLDAECLIP-CMSA)
作图:1.性能对比表格 2.跨模态对齐实验之我设计的注意力机制如何更充分的对齐不同模态的信息以及有着更好的表征,画对应的featuremap。- MEVF1(MAML+DAE-BAN):已做Overcoming Data Limitation in Medical Visual Question Answering?
- MEVF2(pubCLIP+AE-BAN):已做Does CLIP Benefit Visual Question Answering in the Medical Domain as Much as it Does in the General Domain?
- MEVF3(pubCLIP+MAML+AE-BAN):无融合算法,纯concat,如果性能反而下降说明多模态信息中存在特征空间互斥的问题
- OUTMEVF(pubCLIP+MAML+AE-CMSAplus):用了新的方法,得到了性能的提升,证实我的融合方法可以解决上述问题
实验二:CMSA的跨模态表达能力,以及和开放预训练模型CLIP结合后提升的open-end能力
作图:1. 文字图像匹配的注意力图 2. 问答实验- MTCLIP
- NEW DESIGN:混合图像编码器+Bert语言模型+多对齐注意力+贝叶斯决策
实验三:贝叶斯拒绝分类实验,提高模型决策的可解释性和诊断安全性
作图:1. 贝叶斯分类层的输出结果以及其相应的不确定性分布图 2. 拒绝分类实验(拒绝率)- 问题:部分贝叶斯算不算贝叶斯,只在分类层采用贝叶斯有什么意义?
答:根据论文1.《Weight Uncertainty in Neural Network》2.LiBRe: A Practical Bayesian Approach to Adversarial Detection不确定性的由来可知,部分网络的可行性存在,并且起到集成分类器,小样本下抗过拟合的效果 - 问题:为什么加入贝叶斯后性能反而有所上升?但收敛速度不如传统分类器(slake数据集上的实验现象)
答:因为贝叶斯分类器相比传统的分类器计算loss本质是多次采样取均值,并且趋近的是一个变分后验分布。 - OUTMEVF(pubCLIP+MAML+AE-CMSAplus)
- OUTMEVF(pubCLIP+MAML+AE-CMSAplus-Bayes)
- 已经初步验证了参数sigma=0.1 a=0.0001会达到较优水平
实验四:对比学习预训练模型加入Med-VQA的意义
已有实验:一个一个类比实验五:不同融合方法的对比
对比实验
- IDEA
- FRAMWORK
- DATASET
消融实验:证明我方法里某些局部方法的有效性和非敏感性,也就是证明我的思路起了主要作用
- INPT-CMSA:不用特殊预训练过的编码器
- SPTP-CMSA:单任务预训练训练的编码器
- MTPT-BAN:多任务预训练的编码器+双线性池化
- MTPT-CMSA:多任务预训练的编码器+跨模态自注意力
实验结果和测试
CLIP-AE-BAN/SAN
CMSA-MTPT/BAN/SAN
MAML-AE-BAN/SAN
结论
新的视觉模型在图像表达上的提升和原因
不同的编码方式有不同的作用,例如自编码器可以缓解图像噪声问题,元学习模型可以缓解训练样本不足的问题,CLIP对比学习可以解决领域泛化问题,提高开放域问答的能力。如何把握这些特征之间的关联,以及更好地采用这些特征,可以由自注意力机制实现。新的语言模型的改进点和理由
暂无发现注意力模型的改进理由和作用
传统的方法是使用双线性池化方法和注意力堆叠方法,新的方法有交互式注意力不断学习图片的文本的共同表达。贝叶斯推理在此处的意义
重要
参考文献
A Dual-Attention Learning Network with Word and Sentence Embedding for Medical Visual Question Answering
文字材料
融合向量问题和方法的讨论(https://www.zhihu.com/question/359581718):大部分是concat和sum
相比通用领域,医学视觉问答在技术上更具挑战性:
专业知识要求高,专家注释费用昂贵,且无法直接从图像合成问答对。
通用领域图像和医学图像存在差异,基于通用领域图形预训练模型的迁移学习在医疗视觉问答任务上的表现有待提升。
医疗视觉问答需要聚焦在图像的细粒度上,因为病变是微观的。
从现有技术来说,目前的影像分析模块可以被视为一个Close-end的问答系统,回答影像分类的是/否问题,它的问题类型受限、问题领域受限、推理能力有限。未来的分析将会走向Open-End的模式,回答多医学领域、多影像模态的开放式问题。
医疗领域可通过引入外部知识库,如电子病历、知识图谱等,对模型进行优化。另外,医疗领域视觉问答模型的可解释性和可信度是重点问题。在发掘数据规律时,可多采用平衡的数据分布,增加一些正则的模式来缓解单模态下信息偏差的问题。目前来说,深度学习如同一个黑盒,有不可解释性,从建模前、建模中和建模后,透明化一些数据规律,提高模型的可解释性与可信度,也是日后相应研究努力的一个方向。